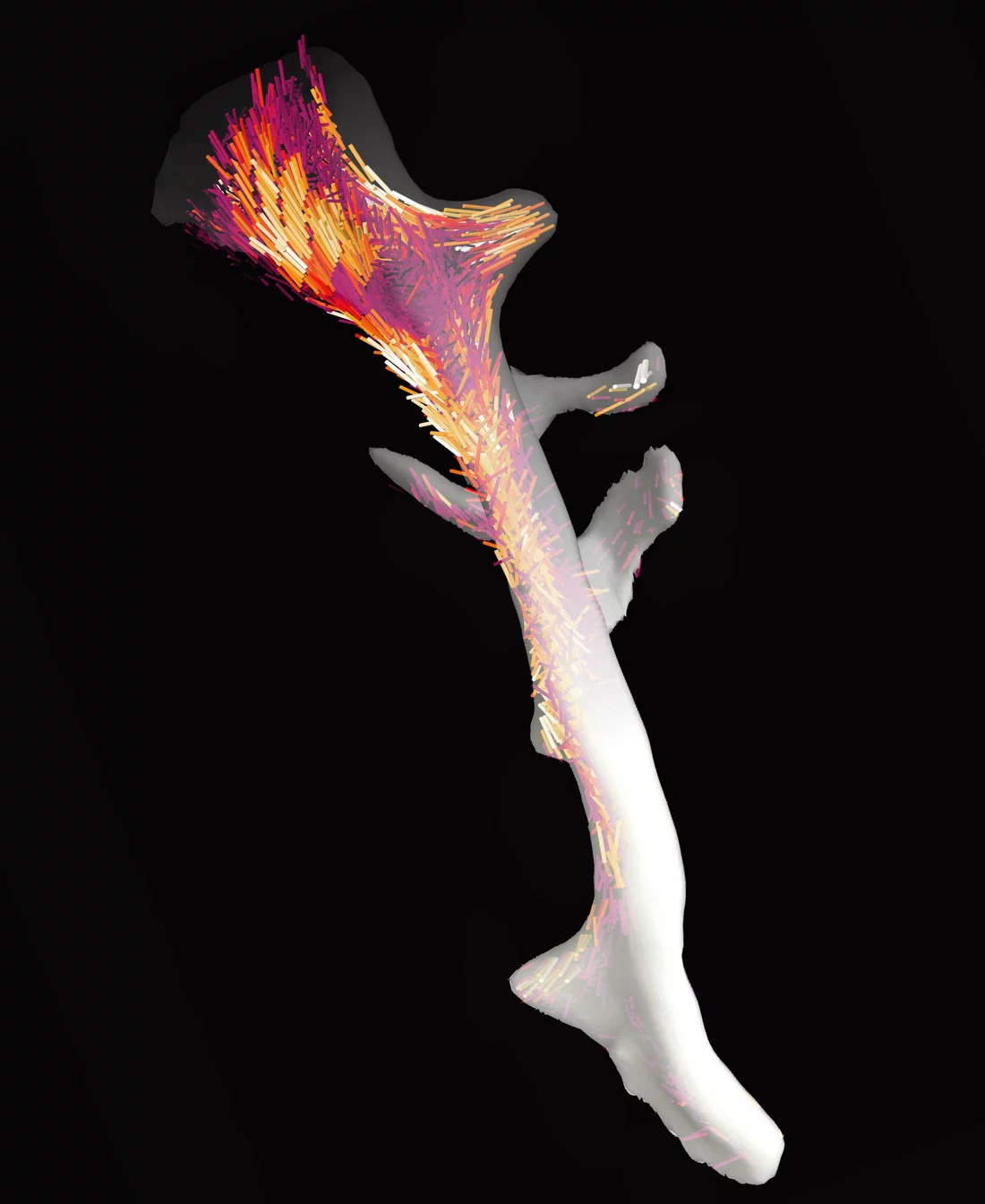
Les os sont composés de minuscules fibres, à peu près mille fois plus fines qu’un cheveu humain, appelées fibrilles de collagène. Une caractéristique essentielle de ces fibrilles de collagène est leur capacité à s’orienter différemment en fonction de la région de l’os à laquelle elles appartiennent. Cette particularité est décisive pour la stabilité mécanique de l’os. Toutefois, la tomographie assistée par ordinateur classique (couramment appelée scanner dans le domaine médical) ne permet de déterminer que la densité des fibrilles, et non leur orientation locale. Des chercheurs de l’Institut Paul Scherrer PSI ont à présent surmonté cette restriction grâce à une méthode d’analyse informatique novatrice. Ils l’ont appliquée à leurs résultats de mesures d’un fragment d’os, effectuées à la Source de Lumière Suisse SLS. Cela leur a permis de déterminer l’agencement local et l’orientation en 3D des fibrilles de collagène à l’intérieur de l’os. A l’avenir, leur méthode pourra aussi être utilisée sur les échantillons biologiques en général, tout comme en sciences des matériaux. Les chercheurs ont publié les résultats de leur étude dans la prestigieuse revue scientifique Nature.
Une nouvelle méthode élaborée par des chercheurs de l’Institut Paul Scherrer PSI permet de visualiser en 3D l’agencement de la nanostructure d’un objet. Les chercheurs en ont fait la démonstration avec un fragment de vertèbre humaine d’une longueur de 2,5 millimètres. Ils ont conduit ces travaux avec des spécialistes de la biomécanique de l’os de l’EPF Zurich et de l’Université de Southampton, en Angleterre. Les os sont constitués de fibres minuscules, appelées fibrilles de collagène, dont on vient de réussir à visualiser l’agencement local dans l’espace et l’orientation sur tout un fragment d’os. Ces propriétés sont déterminantes pour la stabilité de l’os. La technique de visualisation pourrait notamment profiter à la recherche sur l’ostéoporose. Elle se prête de manière générale à l’examen d’échantillons biologiques, mais aussi au développement de matériaux prometteurs.
Les données ont été obtenues à la Source de Lumière Suisse SLS du PSI, où le fragment d’os a été exposé à un faisceau très étroit et très intense de rayons X. Ce faisceau balaie l’échantillon et le mesure point par point. Cela permet de déterminer la nanostructure locale à chaque point de mesure.
L’étape cruciale pour passer de la 2D à la 3D
Jusqu’ici, on ne pouvait balayer et analyser de cette manière que des échantillons bidimensionnels. Le procédé classique consiste à découper en couches très minces l’objet que l’on souhaite étudier. Mais il n’est pas toujours possible de trancher les objets suffisamment finement, rappelle Manuel Guizar-Sicairos, qui a mené le projet. Lors de cette découpe, il arrive même que l’on détruise ou change la nanostructure que l’on voulait justement étudier.
Fondamentalement, mieux vaut aussi opter pour une méthode qui n’endommage pas l’échantillon, avec laquelle l’objet à étudier reste disponible après la réalisation de la mesure.
Pour pouvoir obtenir une visualisation en 3D, les chercheurs du PSI ont balayé leur échantillon à plusieurs reprises, en le tournant d’un petit angle entre deux scans. Ils ont ainsi obtenu des données de mesure à partir de toutes les perspectives dans l’espace, ce qui leur a permis, après coup, de reconstruire l’objet en 3D à l’ordinateur, y compris sa nanostructure.
La nouvelle méthode de mesure des chercheurs du PSI repose sur le principe fondamental de la tomographie assistée par ordinateur (couramment appelée scanner dans le domaine médical). Là aussi, on commence par réaliser de nombreuses radiographies d’un patient ou d’un objet à partir de différentes perspectives, avant de recourir à une analyse informatique pour les assembler de manière à obtenir l’image désirée. Toutefois, le scanner conventionnel n’utilise pas de faisceau étroit de rayons X : il irradie l’objet par superficies.
Le scanner permet de visualiser les variations de densité du matériau, mais pas l’agencement ni l’orientation de la nanostructure fondamentale. Cet objectif ne peut être atteint que par le biais du faisceau étroit et intensif de rayons X de la SLS, et en recourant à des capteurs ultramodernes.
Images générées par algorithmes mathématiques
Toutefois, l’étape la plus complexe a été la recomposition à l’ordinateur d’une image en 3D de l’échantillon à partir d’une quantité colossale de données. Pour ce faire, les chercheurs ont développé eux-mêmes un algorithme mathématique sophistiqué. Le faisceau de rayons X traverse chaque fois toute l’épaisseur de l’échantillon et nous, nous ne voyons que le résultat final, explique Marianne Liebi, première auteure de l’étude. L’aspect de structure tridimensionnelle, nous devons encore le découvrir après coup.
Pour chaque point dans l’échantillon, l’algorithme de Marianne Liebi recherche la structure qui correspond le mieux aux données mesurées. Dans ce cadre, les chercheurs ont exploité le fait suivant : ils pouvaient partir du principe qu’il existe une certaine symétrie au niveau de l’agencement des fibrilles de collagène dans l’os. Cela leur a permis de réduire leurs données à une dimension prévisible. Il restait malgré tout 2,2 millions de paramètres. Ces derniers ont été optimisés par ordinateur, jusqu’à ce que les chercheurs obtiennent l’image de l’échantillon qui expliquait le mieux les mesures obtenues.
Après tant de mathématiques pures, j’ai été surprise de voir apparaître une image qui ressemblait vraiment à un os, avoue Marianne Liebi. Les détails qu’on y découvrait étaient immédiatement évidents.
Comme une carte des zones de végétation
Alors que le scanner classique produit des images avec des niveaux de gris, la nouvelle méthode permet d’obtenir des visualisations presque colorées, avec nettement plus d’informations : les lignes grises indiquent les orientations à l’échelle nanométrique et renseignent même sur l’orientation – autrement dit, elles permettent de savoir si, à un point donné, la majorité des fibrilles de collagène sont alignées parallèlement les unes par rapport aux autres, ou si elles le sont en partie seulement, ou pas du tout.
Nous ne pouvons pas visualiser directement chaque fibrille de collagène, mais nous n’en avons pas besoin non plus, détaille Manuel Guizar-Sicairos. Notre imagerie équivaut plutôt à une carte de zones de végétation : dans ce genre de carte, on décrit des aires et on indique si la zone en question abrite plutôt des conifères, des feuillus ou des forêts mixtes.
Par analogie : les méthodes microscopiques et nanoscopiques conventionnelles nécessitaient cette visualisation des différentes essences d’arbres. D’où la règle qui prévalait jusqu’ici : plus la structure d’un objet était petite, plus le cadrage de l’image devait l’être aussi. Avec leur nouvelle méthode, les chercheurs ont réussi à contourner cette règle : ils ont fixé dans une seule image l’agencement de la nanostructure d’un fragment d’os visible à l’œil nu.
Un second article paraît dans la revue scientifique Nature en même temps que leur publication. Il porte sur des travaux menés sous la houlette d’une autre équipe de recherche, et est également cosignée par Marianne Liebi et Manuel Guizar-Sicairo. Cet article présente une méthode d’analyse alternative, qui aboutit à des résultats de recherche similaires : il s’agit de la nanostructure interne en 3D d’une dent humaine.
Texte : Institut Paul Scherrer/Laura Hennemann
À propos du PSI
L'Institut Paul Scherrer PSI développe, construit et exploite des grandes installations de recherche complexes et les met à la disposition de la communauté scientifique nationale et internationale. Les domaines de recherche de l'institut sont centrés sur la matière et les matériaux, l'énergie et l'environnement ainsi que la santé humaine. La formation des générations futures est un souci central du PSI. Pour cette raison, environ un quart de nos collaborateurs sont des postdocs, des doctorants ou des apprentis. Au total, le PSI emploie 1900 personnes, étant ainsi le plus grand institut de recherche de Suisse. Le budget annuel est d'environ CHF 380 millions.
(Mise à jour : Avril 2015)
Informations supplémentaires
Groupe de recherche pour la diffusion cohérente des rayons X (en anglais)Contact
Dr Marianne Liebi, Groupe de recherche pour la diffusion cohérente des rayons X, Laboratoire de Macromolecules et bio-imagerie, Institut Paul Scherrer,téléphone: +41 56 310 54 53, e-mail: marianne.liebi@psi.ch [allemand, anglais]
Dr Manuel Guizar-Sicairos, Groupe de recherche pour la diffusion cohérente des rayons X, Laboratoire de Macromolecules et bio-imagerie, Institut Paul Scherrer,
téléphone: +41 56 310 34 09, e-mail: manuel.guizar-sicairos@psi.ch [anglais, espagnol]
Publications originales
Nanostructure surveys on macroscopic specimens by small-angle scattering tensor tomographyM. Liebi, M. Georgiadis, A. Menzel, P. Schneider, J. Kohlbrecher, O. Bunk and M. Guizar-Sicairos,
Nature 19 novembre 2015
DOI: 10.1038/nature16056
Six-dimensional real and reciprocal space small-angle X-ray scattering tomography
F. Schaff, M. Bech, P. Zaslansky, C. Jud, M. Liebi, M. Guizar-Sicairos and F. Pfeiffer,
Nature 19 novembre 2015
DOI: 10.1038/nature16060