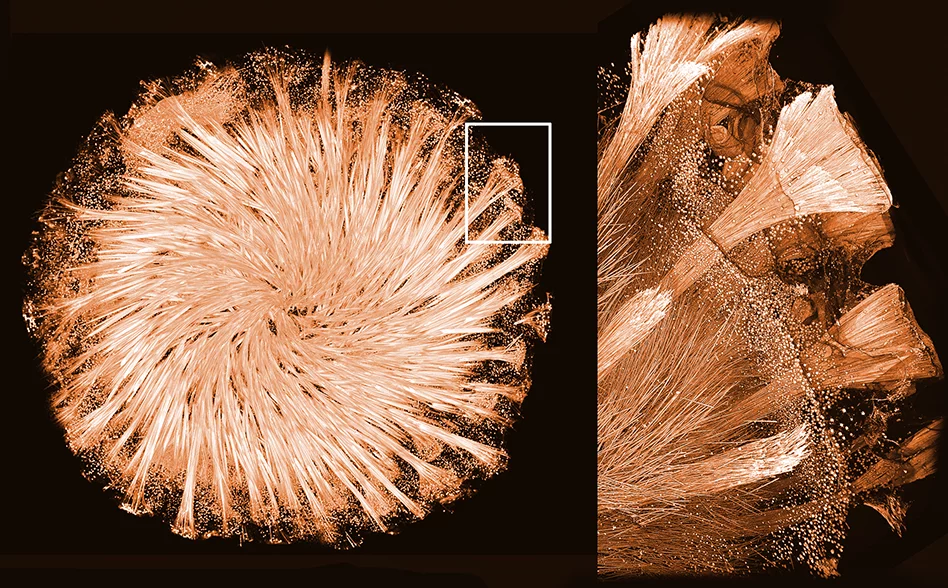
Des mesures menées à la Source de Lumière Suisse SLS ont permis de comprendre la genèse du seul assemblage cristallin hybride minéral-protéine naturel connu à ce jour. Ce cristal fait partie intégrante du fascinant squelette de verre de certaines éponges. Ces créatures, qui comptent parmi les plus anciens animaux que l’on trouve sur Terre, vivent dans de nombreux milieux aquatiques différents, qui vont des lacs aux fonds océaniques. Cela fait longtemps que les scientifiques se demandent comment certaines éponges s’y prennent pour façonner un réseau d’éléments de squelette extrêmement symétriques avec du verre qui, lui, présente une structure désordonné. En collaboration avec l’Institut Paul Scherrer, des chercheurs de l’Université technique de Dresde en Allemagne ont réussi pour la première fois à élucider la structure tridimensionnelle d’une protéine qui est responsable de la formation du verre dans ces éponges. Leurs résultats ont été publiés dans la revue spécialisée Proceedings of the National Academy of Sciences of the USA.
Comme leur nom l’indique, les éponges de verre – ou éponges de silicate – ont un squelette à base de verre, constitué d’un réseau d’aiguilles, de crochets, d’étoiles et de sphères. Pour réaliser une architecture aussi unique, ces éponges doivent manipuler du verre pour façonner des éléments extrêmement réguliers et symétriques. Or ce verre présente, lui, une structure désordonnée.
Les canaux qui se trouvent à l’intérieur de ces éléments de verre contiennent de fines fibres cristallines, qui sont constituées d’une protéine dénommée silicatéine. On sait que les cristaux de silicatéine sont responsables de la synthèse du verre dans les éponges et de la mise en forme du squelette de verre. Les tentatives menées jusqu’ici pour élucider la structure 3D de cette protéine, afin de décrire comment elle s’assemble en cristaux et comment ceux-ci forment le squelette de verre, avaient échoué. Notamment parce que personne n’était en mesure de cultiver ces cristaux en laboratoire.
Une équipe de chercheurs emmenée par Igor Zlotnikov, du B CUBE - Center for Molecular Bioengineering à l’Université technique de Dresde, a opté pour une approche différente: au lieu de produire de la silicatéine en laboratoire, les scientifiques ont décidé de prendre les aiguilles de verre d’un squelette d’éponge et d’analyser les minuscules cristaux qu’elles contenaient déjà. Le problème: ces cristaux étaient si minuscules qu’il a fallu recourir à quelques astuces. C’est là qu’une équipe de la Source de Lumière Suisse SLS au PSI est venue en renfort.
Beaucoup de petits cristaux au lieu d’un grand
Une méthode traditionnelle d’élucidation de la structure 3D d’une protéine consiste à exposer son cristal à de la lumière de type rayons X. Dans un cristal de protéine, les atomes diffusent la lumière de type rayons X et fournissent ainsi un instantané unique de son agencement interne. En imprimant une rotation au cristal et en collectant ces instantanés réalisés sous de nombreux angles, les chercheurs obtiennent suffisamment de données pour décrypter la structure 3D de la protéine, par recours à certaines méthodes de calcul.
Cette méthode largement utilisée fonctionne bien pour des cristaux d’au moins 10 micromètres. Cependant, le groupe d’Igor Zlotnikov souhaitait analyser des cristaux de silicatéine environ dix fois plus petits. Or lorsque ces derniers étaient exposés aux rayons X, ils étaient presque immédiatement endommagés, ce qui rendait impossible la collecte d’un jeu complet de données obtenues à partir de différents angles.
Avec le soutien de l’équipe de la Source de Lumière Suisse SLS du PSI, les chercheurs ont donc utilisé une nouvelle méthode, la cristallographie sérielle. «Cette méthode consiste à combiner des diagrammes de diffraction de nombreux cristaux, explique Filip Leonarski, scientifique spécialiste des lignes de faisceau au PSI, qui a participé à l’étude. Avec la méthode traditionnelle, vous tournez un film, pour ainsi dire. Avec la nouvelle méthode, vous obtenez de nombreux instantanés que vous combinez ensuite pour décrypter la structure.» Chaque instantané est réalisé sur une partie différente de ces minuscules cristaux, voire sur un autre cristal.
En tout, les chercheurs ont collecté plus de 3500 clichés de diffraction des rayons X de 90 aiguilles de verre, avec des orientations complètement aléatoires. Des méthodes de calcul ultramodernes leur ont permis d’identifier de l’ordre dans le chaos et d’assembler les données pour élucider la première structure 3D complète de la silicatéine. «L’analyse des données s’est avérée quelque peu fastidieuse, relève Filip Leonarski. Par chance, nous avons déjà beaucoup d’expérience avec cette méthode, car nous y recourons de manière intensive au laser à rayons X à électrons libres SwissFEL.»
Superstructure verre-protéine
La structure 3D de la silicatéine qu’ils ont obtenue a permis aux chercheurs de comprendre la structure et la fonction de cette protéine au sein du squelette de verre de l’éponge. Le groupe d’Igor Zlotnikov au Center for Advancing Electronics de Dresde (cfaed) a par ailleurs recouru à la microscopie électronique en transmission à haute résolution (HRTEM) pour examiner plus précisément les cristaux de silicatéine contenus dans les aiguilles de verre.
«Nous avons observé une structure exceptionnellement ordonnée et en même temps complexe, explique Igor Zlotnikov. En analysant l’échantillon, nous avons constaté qu’il s’agissait d’un mélange de matière organique et inorganique. Cela signifie que les protéines et le verre forment une superstructure hybride qui façonne en quelque sorte le squelette des éponges.» Igor Zlotnikov qualifie cette structure de superstructure protéine-verre fonctionnelle 3D, décrite pour la première fois de manière détaillée dans un organisme vivant.
Texte: basé sur un communiqué de presse de l’Université technique de Dresde avec des compléments de l’Institut Paul Scherrer/Brigitte Osterath
Lien: https://tu-dresden.de/tu-dresden/newsportal/news/ordnung-im-chaos-finde…
Contact
Dr Filip Leonarski
Laboratoire de Macromolécules et de bio-imagerie
Institut Paul Scherrer, Forschungsstrasse 111, 5232 Villigen PSI, Suisse
Téléphone: +41 56 310 56 52, e-mail: filip.leonarski@psi.ch [anglais, polonais]
Dr Igor Zlotnikov
B CUBE - Center for Molecular Bioengineering
Université technique de Dresde, Tatzberg 41, 01307 Dresde, Allemagne
Téléphone: +49 351 463 43090, e-mail: igor.zlotnikov@tu-dresden.de
Publication originale
Natural hybrid silica/protein superstructure at atomic resolution
S. Görlich, A.J. Samuel, R.J. Best, R. Seidel, J. Vacelet, F.K. Leonarski, T. Tomizaki, B. Rellinghaus, D. Pohl, I. Zlotnikov
Proceedings of the National Academy of Sciences of the USA, 23 novembre 2020 (en ligne)
DOI: 10.1073/pnas.2019140117
Droit à l'utilisation
Le PSI fournit gratuitement des images et/ou du matériel vidéo pour la couverture médiatique du contenu du texte ci-dessus. L'utilisation de ce matériel à d'autres fins n'est pas autorisée. Cela inclut également le transfert des images et du matériel vidéo dans des bases de données ainsi que la vente par des tiers.